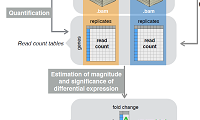
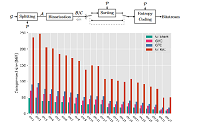
In den letzten Jahren haben technologische Fortschritte bei der DNA-Sequenzierung zu schnelleren und kostengünstigeren Ansätzen für die Sequenzierung individueller Genome und anderer genomischer Proben geführt. Aufgrund der enormen Menge der erzeugten Sequenzierungsdaten stellt die Verarbeitung, Speicherung und Analyse von Sequenzierungsdaten die wissenschaftliche Gemeinschaft vor neue Herausforderungen. Es müssen neue Methoden und Werkzeuge entwickelt werden, um das Verständnis der zugrundeliegenden Biologie zu verbessern und die derzeitigen Einschränkungen in Bezug auf Speicherplatz, Verarbeitungsgeschwindigkeit und vieles mehr zu überwinden.